Dynamique conformationelle de peptides et protéines
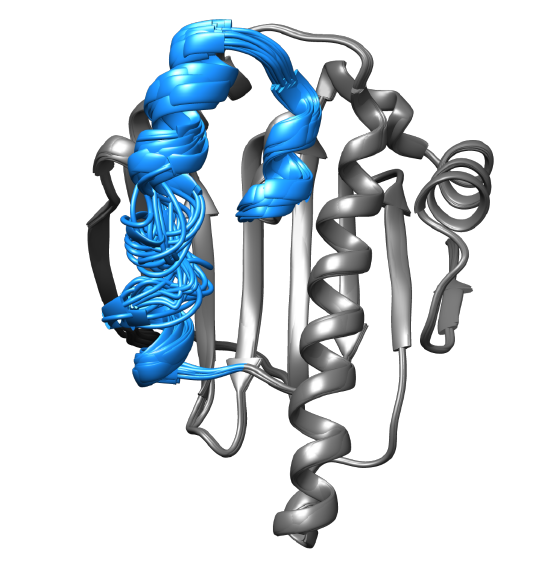
De nombreuses protéines et peptides associent la dynamique conformationnelle à la fonction biologique et présentent des hétérogénéités structurelles dans des conditions de solution normales. Les mouvements dans les biomolécules sont complexes car ils comportent de nombreuses contributions de mouvements avec des mécanismes différents, se produisant à des échelles de durée et de temps diverses. Pour les comprendre, différentes techniques doivent être combinées. En collaboration avec des expérimentateurs spécialisés dans les expériences de RMN, nous étudions la dynamique conformationnelle de différentes protéines en utilisant des simulations de dynamique moléculaire et des techniques d'analyse de pointe. Nous nous intéressons particulièrement aux peptides imitant le collagène, aux isomérases et aux chaperons, ainsi qu'aux mécanismes d'allostère dynamique qui s'y déroulent.
D'autre part, nous nous étudions les interactions moléculaire entre différentes protéines et les polluants PFAS (per- and poly-fluoro alkyl substances) grâce des techniques de docking et de dynamique moléculaire biaisée.
Collaborations :Laboratoire des Biomolécules, Paris; Institut de Biologie Structurale, Grenoble; Matrice Extracellulaire et Dynamique Cellulaire MEDYC, Reims
Financements :ANR CH2PROBE; Région Auvergne-Rhône-Alpes DYN4DRUGS; MITI : pollutions et dépollutions
Conformational Dynamics of Peptides and Proteins

Many proteins and peptides link conformational dynamics to biological function and exhibit structural heterogeneities under normal solution conditions. Motions in biomolecules are complex because they involve numerous contributions from movements with different mechanisms, occurring over a wide range of length and time scales. To understand them, different techniques must be combined. In collaboration with researchers specializing in NMR experiments, we study the conformational dynamics of various proteins using molecular dynamics simulations and advanced analysis techniques. We are particularly interested in collagen-mimicking peptides, isomerases, and chaperones, as well as the dynamic allosteric mechanisms that take place within them.
On the other hand, we also study the molecular interactions between different proteins and PFAS pollutants (per- and polyfluoroalkyl substances) using docking methods and biased molecular dynamics simulations.
Collaborations :Laboratoire des Biomolécules, Paris; Institut de Biologie Structurale, Grenoble; Matrice Extracellulaire et Dynamique Cellulaire MEDYC, Reims
Fundings:ANR CH2PROBE; Région Auvergne-Rhône-Alpes DYN4DRUGS; MITI : pollutions et dépollutions