Dynamique conformationelle de peptides et protéines
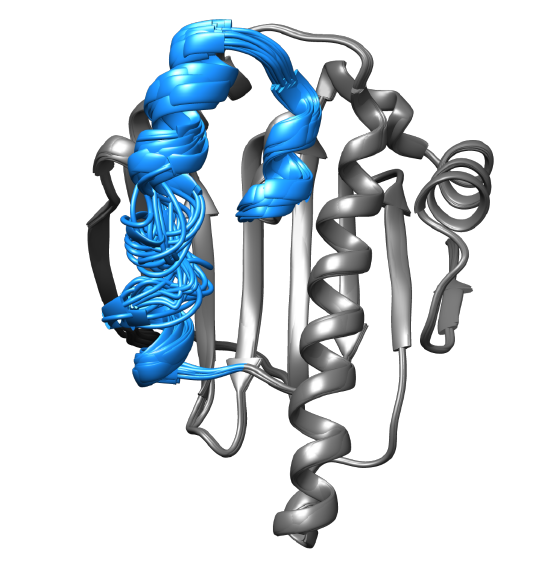
De nombreuses protéines et peptides associent la dynamique conformationnelle à la fonction biologique et présentent des hétérogénéités structurelles dans des conditions de solution normales. Les mouvements dans les biomolécules sont complexes car ils comportent de nombreuses contributions de mouvements avec des mécanismes différents, se produisant à des échelles de durée et de temps diverses. Pour les comprendre, différentes techniques doivent être combinées. En collaboration avec des expérimentateurs spécialisés dans les expériences de RMN, nous étudions la dynamique conformationnelle de différentes protéines en utilisant des simulations de dynamique moléculaire et des techniques d'analyse de pointe. Nous nous intéressons particulièrement aux peptides imitant le collagène, aux isomérases et aux chaperons, ainsi qu'aux mécanismes d'allostère dynamique qui s'y déroulent.
Collaborations :Laboratoire des Biomolécules, Paris; Institut de Biologie Structurale, Grenoble
Financements :ANR CH2PROBE; Région Auvergne-Rhône-Alpes DYN4DRUGS
Conformational Dynamics of Peptides and Proteins

Supported lipid layers are composed of ordered lipids covering the surface of a solid which are commonly used to study biomolecular phenomena, e.g. protein and peptide adsorptions. One of the advantages of these model systems is that they can be synthesized with a precise control of their composition. In collaboration with experimentalists, we aim at studying the mechanical properties of stacks of lipid membranes, which play a role in reducing friction in living bodies. The understanding of the microscopic mechanisms involved in biolubrication remains very phenomenological, mainly due to the structural complexity of these systems. Moreover, the hydrodynamics of the complex lipidic fluid confined by the substrate is not yet fully understood. Experimental partners investigate highly controlled supported lipidic layers under mechanical stress, using recent instrumental development, allowing to study both the tribology and velocity profile of confined systems. One aim is to compare the microscopic quantities measured with numerical simulations. At ILM, we develop the numerical simulations of the complete system under mechanical stress : substrate, solvent, lipid layers and seek to answer the following questions: What are the correlations between structure and tribology ? What is the role of the hydrodynamics of the confined water and counterions layers ?
Collaborations :Laboratoire des Biomolécules, Paris; Institut de Biologie Structurale, Grenoble
Fundings:ANR CH2PROBE; Région Auvergne-Rhône-Alpes DYN4DRUGS