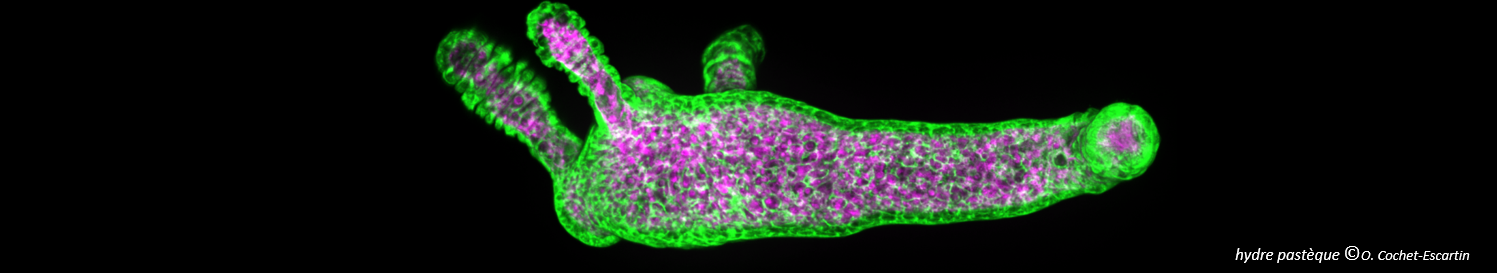
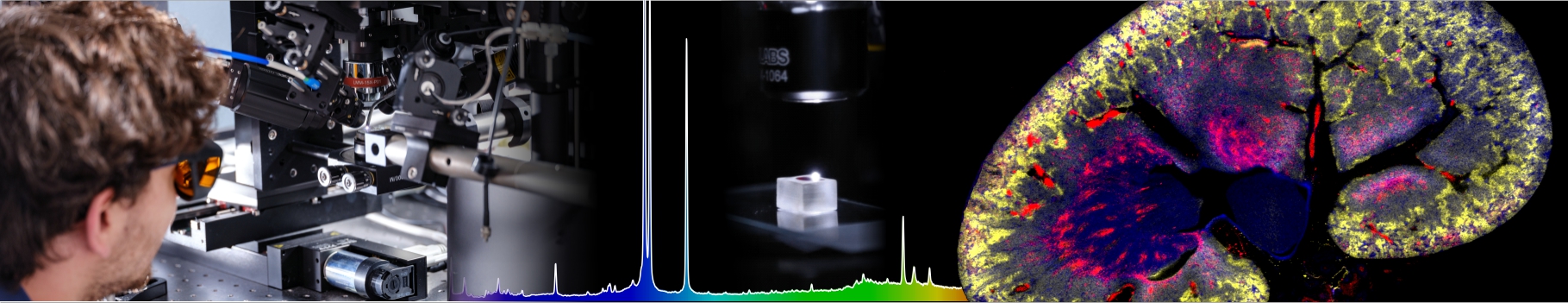
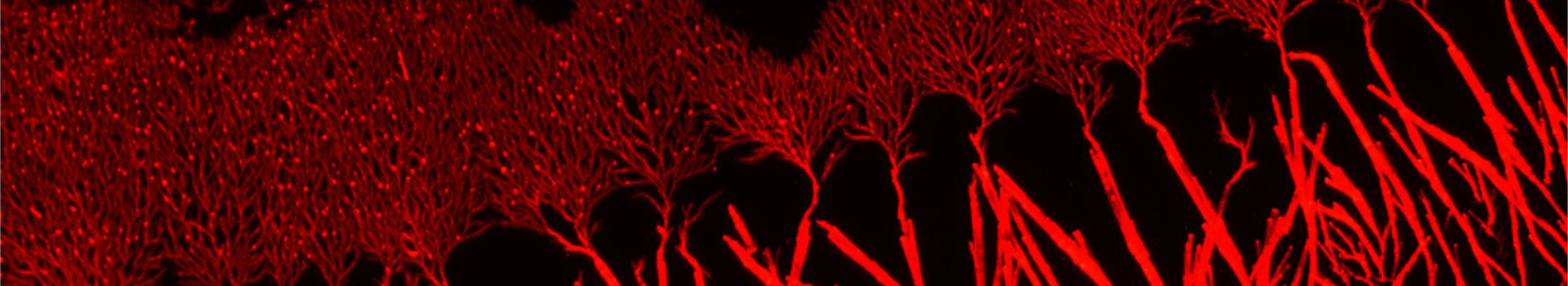
Fibrils or not fibrils...
 Steve Daly, Alex Kulesza et leurs collègues du groupe de P. Dugourd (équipe “Spectrobio”) ont publié un article intitulé "Conformational changes in Amyloid-Beta (12-28) alloforms studied using action-FRET, IMS and molecular dynamics simulations." dans la revue Chemical Science.
Steve Daly, Alex Kulesza et leurs collègues du groupe de P. Dugourd (équipe “Spectrobio”) ont publié un article intitulé "Conformational changes in Amyloid-Beta (12-28) alloforms studied using action-FRET, IMS and molecular dynamics simulations." dans la revue Chemical Science.
 Dans la maladie d’Alzheimer, de petits agrégats de la protéine β-Amyloïde jouent le rôle d’agents infectieux. Mais il semble que des mutations dans un petit cœur hydrophobe formé par les acides aminés 17-21 altèrent considérablement la capacité de cette protéine à former des fibrilles de grande taille. Pour comprendre l’origine de cette altération, les familles de conformation adoptées par la protéine mutée et non mutée ont été comparées : soumises à des tensions, les deux protéines se déforment et perdent progressivement leur structure initiale. En revanche, lors de ce processus de déstructuration, seule la protéine « sauvage » (non mutée) passe par un intermédiaire stable contenant les structures secondaires en feuillets β impliquées dans les fibrilles pathologiques. Pour parvenir à ces résultats, les techniques développées dans le groupe de P.Dugourd : FRET d’action et mobilité ionique ont été combinées à des simulations de dynamique moléculaire.
Dans la maladie d’Alzheimer, de petits agrégats de la protéine β-Amyloïde jouent le rôle d’agents infectieux. Mais il semble que des mutations dans un petit cœur hydrophobe formé par les acides aminés 17-21 altèrent considérablement la capacité de cette protéine à former des fibrilles de grande taille. Pour comprendre l’origine de cette altération, les familles de conformation adoptées par la protéine mutée et non mutée ont été comparées : soumises à des tensions, les deux protéines se déforment et perdent progressivement leur structure initiale. En revanche, lors de ce processus de déstructuration, seule la protéine « sauvage » (non mutée) passe par un intermédiaire stable contenant les structures secondaires en feuillets β impliquées dans les fibrilles pathologiques. Pour parvenir à ces résultats, les techniques développées dans le groupe de P.Dugourd : FRET d’action et mobilité ionique ont été combinées à des simulations de dynamique moléculaire.